生存资料决策曲线分析DCA |
您所在的位置:网站首页 › 随机森林 生存分析 › 生存资料决策曲线分析DCA |
生存资料决策曲线分析DCA
|
本文首发于公众号:医学和生信笔记 “医学和生信笔记,专注R语言在临床医学中的使用,R语言数据分析和可视化。主要分享R语言做医学统计学、meta分析、网络药理学、临床预测模型、机器学习、生物信息学等。 前面介绍了logistic回归的DCA的5种绘制方法,今天学习下cox回归的DCA绘制方法。也是有多种方法可以实现,但我比较推荐能返回数据,用ggplot2自己画的那种。 生存资料的DCA 方法1方法2方法3方法4 生存资料的DCA 方法1使用dcurves包,使用的数据集是包自带的df_surv数据集,一共有750行,9列,其中ttcancer是时间,cancer是结局事件,TRUE代表有癌症,FALSE代表没有癌症。 并不是只有结局事件是生存或者死亡的才叫生存资料哦!只要是time-event类型的,都可以。 # 加载R包和数据,不知道怎么安装的请看我前面的推文library(dcurves)library(survival)data("df_surv")# 查看数据结构dim(df_surv) ## [1] 750 9 str(df_surv) ## tibble [750 × 9] (S3: tbl_df/tbl/data.frame)## $ patientid : num [1:750] 1 2 3 4 5 6 7 8 9 10 ...## $ cancer : logi [1:750] FALSE FALSE FALSE FALSE FALSE FALSE ...## $ ttcancer : num [1:750] 3.009 0.249 1.59 3.457 3.329 ...## $ risk_group : chr [1:750] "low" "high" "low" "low" ...## $ age : num [1:750] 64 78.5 64.1 58.5 64 ...## $ famhistory : num [1:750] 0 0 0 0 0 0 0 0 0 0 ...## $ marker : num [1:750] 0.7763 0.2671 0.1696 0.024 0.0709 ...## $ cancerpredmarker: num [1:750] 0.0372 0.57891 0.02155 0.00391 0.01879 ...## $ cancer_cr : Factor w/ 3 levels "censor","diagnosed with cancer",..: 1 1 1 1 1 1 1 2 1 1 ...这个包使用起来很别扭,但是可以说它很灵活! 如果预测变量只有1个,且是0,1表示的,那就很简单,直接用就行;如果有多个预测变量,就需要先计算出预测概率,然后才能使用。 预测变量是famhistory,这是0,1表示的二分类变量: library(ggplot2)dcurves::dca(Surv(ttcancer, cancer) ~ famhistory, data = df_surv, time = 1 # 时间选1年 ) %>% plot(smooth = T)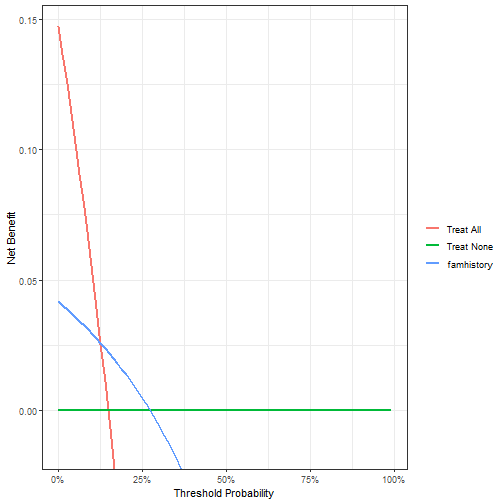 unnamed-chunk-2-146299048
unnamed-chunk-2-146299048
下面展示一个把多个模型的DCA画在一起的例子,和之前介绍的dca.r的用法优点类似。 cancerpredmarker这一列已经是概率了,marker是数值型的连续性变量,famhistory是0,1表示的二分类变量。 dcurves::dca(Surv(ttcancer, cancer) ~ cancerpredmarker + marker + famhistory, data = df_surv, as_probability = "marker", # 只有marker需要转换成概率 time = 1, label = list(cancerpredmarker = "Prediction Model", marker = "Biomarker")) %>% plot(smooth = TRUE,show_ggplot_code = T) + ggplot2::labs(x = "Treatment Threshold Probability") ## # ggplot2 code to create DCA figure -------------------------------## as_tibble(x) %>%## dplyr::filter(!is.na(net_benefit)) %>%## ggplot(aes(x = threshold, y = net_benefit, color = label)) +## stat_smooth(method = "loess", se = FALSE, formula = "y ~ x", ## span = 0.2) +## coord_cartesian(ylim = c(-0.0147287067742928, 0.147287067742928## )) +## scale_x_continuous(labels = scales::percent_format(accuracy = 1)) +## labs(x = "Threshold Probability", y = "Net Benefit", color = "") +## theme_bw()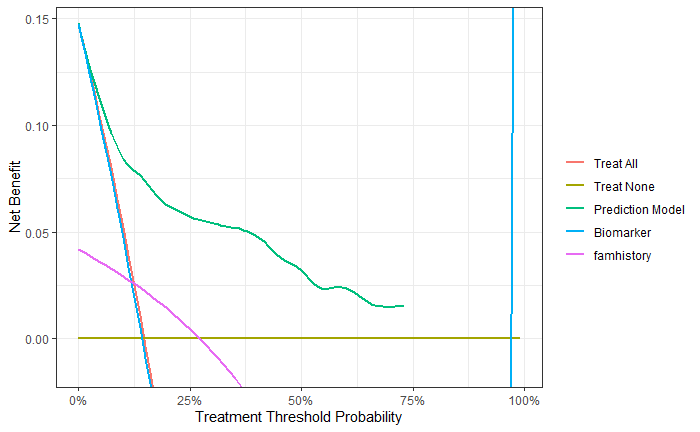 image-20220620205335965
image-20220620205335965
可以看到marker这个曲线有点过分了。。结果也给出了ggplot2的代码,大家可以自己修改。 上面是多个模型在同一个时间点的DCA曲线,如果是同一个模型在不同时间点的DCA,这个包不能直接画出,需要自己整理数据,因为不同时间点进行治疗的风险和获益都是不一样的,所以会出现同一个阈值概率对应多个净获益的情况,所以none和all每个概率阈值下都有1套数据。 如果你的预测变量是多个,就需要先计算预测概率。 # 构建一个多元cox回归cox_model |
【本文地址】
今日新闻 |
推荐新闻 |